一、Aumenta a sensibilità di u sistema di reazione:
1. Isolate RNA di alta qualità:
A sintesi di cDNA successu vene da RNA di alta qualità.L'ARN d'alta qualità deve esse almenu à longu andà è senza inhibitori di transcriptase inversa cum'è EDTA o SDS.A qualità di l'RNA determina a quantità massima di informazioni di sequenza chì pudete trascrive in cDNA.Un metudu cumuni di purificazione di l'RNA hè un metudu in un passu cù guanidine isothiocyanate / acid phenol.Per prevene a contaminazione da tracce di RNase, RNA isolatu da campioni ricchi di RNase (cum'è pancreas) deve esse guardatu in formaldeide per priservà RNA d'alta qualità, soprattuttu per u almacenamentu longu.L'RNA estratto da u fegato di u ratu hè statu basamente degradatu dopu esse almacenatu in l'acqua per una settimana, mentre chì l'RNA estratto da u spleen di u ratu hè statu stabile dopu esse almacenatu in acqua per 3 anni.Inoltre, i trascrizioni più longu di 4 kb sò più sensibili à a degradazione da trace RNases cà i trascrizioni chjuchi.Per aumentà a stabilità di i campioni di RNA almacenati, l'RNA pò esse dissolutu in formamide deionized è almacenatu à -70 ° C.A formamide utilizata per priservà l'RNA deve esse libera di detriti degradanti di l'ARN.L'RNA da u pancreas pò esse cunservatu in formamide per almenu un annu.Quandu si preparanu à utilizà RNA, pudete aduprà u metudu seguente per precipitate RNA: aghjunghje NaCl à 0.2M è 4 volte u voluminu di etanol, mette à a temperatura di l'ambienti per 3-5 minuti, è centrifuga à 10 000 × g per 5 minuti.
2. Aduprate a transcriptase inversa RNaseH-inattiva (RNaseH-) :
L'inhibitori di RNase sò spessu aghjuntu à e reazzioni di trascrizione inversa per aumentà a durata è u rendiment di a sintesi di cDNA.L'inhibitori di RNase deve esse aghjuntu durante a reazione di sintesi di a prima fila in presenza di un buffer è un agentu riduzzione (cum'è DTT), perchè u prucessu prima di a sintesi di cDNA denatura l'inhibitore, liberando cusì RNase ligata chì pò degrada l'RNA.L'inhibitori di a proteina RNase impediscenu solu a degradazione di l'RNA da RNase A, B, C, è ùn impediscenu micca a RNase nantu à a pelle, cusì attentu à ùn intruduce RNase da i vostri ditte malgradu l'usu di sti inhibitori.
A transcriptase inversa catalizza a cunversione di RNA in cDNA.Sia M-MLV è AMV anu attività RNaseH endogena in più di a so propria attività di polimerasi.L'attività di RNaseH è l'attività di polimerasi cumpetenu l'una cù l'altru per u filamentu hibridu furmatu trà u mudellu di RNA è u filamentu di l'ADN primer o l'estensione di cDNA, è degradanu u filamentu di RNA in u cumplessu RNA:DNA.U mudellu di RNA degradatu da l'attività RNaseH ùn pò più serve com'è un sustrato efficace per a sintesi di cDNA, chì riduce u rendiment è a durata di a sintesi di cDNA.Dunque, saria benefiziu per eliminà o riduce assai l'attività RNaseH di a transcriptase inversa.。
SuperScript Ⅱ reverse transcriptase, RNaseH- MMLV reverse transcriptase è thermoScript reverse transcriptase, RNaseH-AMV, ponu ottene più quantità è più cDNA full-length cà MMLV è AMV.A sensibilità RT-PCR serà affettata da a quantità di sintesi di cDNA.ThermoScript hè assai più sensibile cà AMV.A dimensione di i prudutti RT-PCR hè limitata da l'abilità di a transcriptase inversa per sintetizà cDNA, soprattuttu quandu clone cDNA più grande.In cunfrontu cù MMLV, SuperScripⅡ hà aumentatu significativamente u rendiment di i prudutti RT-PCR longu.A transcriptase inversa RNaseH hà ancu aumentatu a termostabilità, perchè a reazione pò esse realizata à temperature più altu ch'è u normale 37-42 ° C.In e cundizioni di sintesi suggerite, utilizate primer oligo(dT) è 10 μCi di [α-P]dCTP.U rendiment tutale di u primu brin hè statu calculatu cù u metudu di precipitazione TCA.L'ADNc di lunghezza completa hè statu analizatu utilizendu bande di dimensioni classificate escise è cuntate nantu à un gel d'agarose alkaline.
3. Aumentà a temperatura di incubazione per a trascrizione inversa:
Una temperatura d'incubazione più alta aiuta à apre a struttura secundaria di RNA, aumentendu u rendiment di a reazione.Per a maiò parte di i mudelli di RNA, l'incubazione di l'RNA è di i primers à 65 ° C senza buffer o sali, seguita da un rapidu raffreddamentu nantu à u ghjacciu eliminerà a maiò parte di e strutture secondarie è permette à i primers di ligà.In ogni casu, certi mudelli anu sempre strutture secondarie, ancu dopu a denaturazione di u calore.L'amplificazione di sti mudelli difficiuli pò esse realizatu utilizendu ThermoScript Reverse Transcriptase è mette a reazione di trascrizzione inversa à una temperatura più altu per migliurà l'amplificazione.Temperature d'incubazione più elevate ponu ancu aumentà a specificità, soprattuttu quandu i primers gene-specifici (GSP) sò usati per a sintesi di cDNA (vede u Capitu 3).Si l'on utilise GSP, assurez-vous que la Tm des amorces soit la même que la température d'incubation attendue.Ùn aduprate micca oligo (dT) è primers aleatoriu sopra à 60 ° C.I primers casuali necessitanu incubazione à 25 ° C per 10 minuti prima di cresce à 60 ° C.In più di utilizà una temperatura di trascrizione inversa più alta, a specificità pò ancu esse migliurata trasferendu direttamente u mischju di RNA/primer da a temperatura di denaturazione di 65 ° C à a temperatura d'incubazione di trascrizzione inversa è aghjunghjendu una mistura di reazione 2x pre-riscaldata (sintesi hot-start cDNA).Stu approcciu aiuta à prevene l'accoppiamentu di basa intermolecular chì si trova à a temperatura più bassa.U cambiamentu di temperatura multiplici necessariu per RT-PCR pò esse simplificatu cù un termociclatore.
Tth polimerasi termostabile agisce cum'è DNA polimerasi in presenza di Mg2+ è cum'è RNA polimerasi in presenza di Mn2+.Pò esse tenutu caldu à una temperatura massima di 65 ° C.In ogni casu, a prisenza di Mn2 + durante a PCR riduce a fideltà, chì rende a polimerasi Tth menu adattata per l'amplificazione d'alta precisione, cum'è a clonazione di cDNA.Inoltre, Tth hà una bassa efficienza di trascrizzione inversa, chì reduce a sensibilità, è, postu chì a trascrizione inversa è a PCR ponu esse realizate cù una sola enzima, e reazzioni di cuntrollu senza trascrizzione inversa ùn ponu esse usate per paragunà i prudutti di amplificazione di cDNA cù DNA genomicu contaminante.I prudutti di amplificazione sò stati separati.
4. Additivi chì prumove a trascrizione inversa:
Additivi cumpresi glicerol è DMSO sò aghjuntu à a reazione di sintesi di u primu filu, chì ponu riduce l'stabilità di l'acidu nucleicu à doppia fila è untie a struttura secundaria di RNA.Finu à 20% glicerol o 10% DMSO pò esse aghjuntu senza affettà l'attività SuperScript II o MMLV.AMV pò ancu tollerà finu à u 20% di glicerol senza perdita di attività.Per maximizà a sensibilità di RT-PCR in a reazione di trascrizzione inversa SuperScriptⅡ, 10% di glicerol pò esse aghjuntu è incubatu à 45 ° C.Se 1/10 di u pruduttu di reazzione di trascrizzione inversa hè aghjuntu à a PCR, allora a cuncentrazione di glicerol in a reazione di amplificazione hè di 0,4%, chì ùn hè micca abbastanza per inibisce a PCR.
5. Trattamentu di RNaseH:
U trattamentu di e reazzioni di sintesi di cDNA cù RNaseH prima di a PCR pò aumentà a sensibilità.Per certi mudelli, hè pensatu chì l'RNA in a reazione di sintesi di cDNA impedisce u ligame di i prudutti di amplificazione, in quale casu u trattamentu RNaseH pò aumentà a sensibilità.In generale, u trattamentu RNaseH hè necessariu quandu si amplificanu mudelli di destinazione di cDNA più longu, cum'è a scherosi tuberosa II di bassa copia.Per questu mudellu difficiuli, u trattamentu RNaseH hà rinfurzatu u signale pruduciutu da SuperScript II o cDNA sintetizatu da AMV.Per a maiò parte di e reazioni RT-PCR, u trattamentu RNaseH hè opzionale, perchè u passu di denaturazione PCR à 95 ° C generalmente idrolizza l'RNA in u cumplessu RNA:DNA.
6. Migliuramentu di Small RNA Detection Method:
RT-PCR hè soprattuttu sfida quandu solu picculi quantità di RNA sò dispunibili.U glucogenu aghjuntu cum'è un trasportatore durante l'isolamentu di RNA aiuta à aumentà u rendiment di picculi campioni.U glucogenu senza RNase pò esse aghjuntu à u stessu tempu chì aghjunghje Trizol.U glucogenu hè soluble in acqua è pò esse mantinutu in a fase aquosa cù RNA per aiutà a precipitazione successiva.Per campioni di menu di 50 mg di tissutu o 106 cellule cultivate, a cuncentrazione cunsigliata di glicogenu senza RNasi hè 250 μg/ml.
Aghjunghjendu BSA acetilatu à a reazione di trascrizzione inversa cù SuperScript II pò aumentà a sensibilità, è per picculi quantità di RNA, riducendu a quantità di SuperScript II è aghjunghjendu 40 unità di RNaseOut nuclease inhibitor pò aumentà u livellu di deteczione.Se u glicogenu hè utilizatu in u prucessu di isolamentu di RNA, hè sempre cunsigliatu di aghjunghje BSA o RNase inhibitor quandu si usa SuperScript II per a reazione di trascrizione inversa.
二、Aumenta a specificità RT-PCR
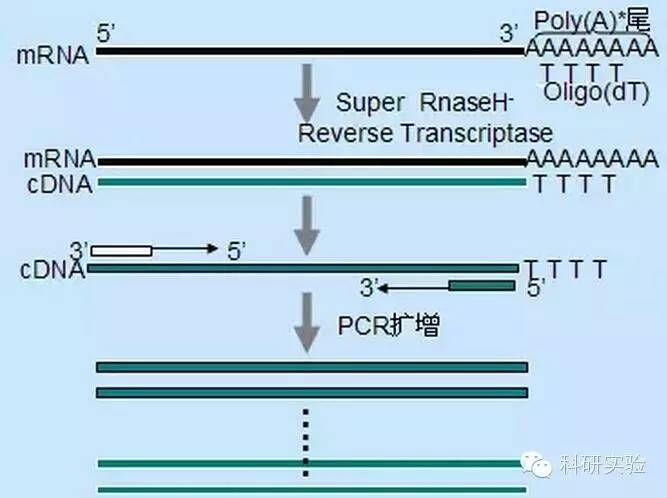
1. Asintesi CND:
A sintesi di cDNA di u primu filu pò esse iniziata cù trè metudi diffirenti, a specificità relativa di quale afecta a quantità è u tipu di cDNA sintetizatu.
U metudu di prima aleatoriu era u menu specificu di i trè metudi.I primers anneal in parechji siti in tutta a trascrizione, generando cDNA brevi, di lunghezza parziale.Stu metudu hè spessu usatu per ottene sequenze finali 5′ è per ottene cDNA da mudelli di RNA cù regioni di struttura secundaria o cù siti di terminazione chì ùn ponu micca esse replicati da a transcriptase inversa.Per ottene u cDNA più longu, u rapportu di primers à RNA in ogni mostra di RNA deve esse determinatu empiricamente.La concentrazione iniziale di primers casuali variava da 50 a 250 ng per 20 μl di reazione.Siccomu l'ADNc sintetizatu da l'RNA tutale cù primers aleatoriu hè principalmente RNA ribosomi, u poli (A) + RNA hè generalmente sceltu cum'è mudellu.
I primers Oligo (dT) sò più specifichi cà i primers random.Si ibrida à a coda poli (A) chì si trova à l'estremità 3' di a maiò parte di l'ARNm eucarioti.Perchè u poli (A) + RNA hè di circa 1% à 2% di l'RNA tutale, a quantità è a cumplessità di cDNA hè assai menu cà cù primers aleatoriu.A causa di a so alta specificità, l'oligo (dT) generalmente ùn necessita micca ottimisazione di u rapportu di RNA à primers è poly (A) + selezzione.Hè cunsigliatu di utilizà 0.5μg oligo (dT) per 20μl sistema di reazione.oligo (dT) 12-18 hè adattatu per a maiò parte di RT-PCR.U Sistema ThermoScript RT-PCR offre oligo (dT) 20 per via di a so megliu stabilità termica per temperature d'incubazione più elevate.
Gene specific primers (GSP) sò i primers più specifichi per u passu di trascrizione inversa.GSP hè un oligonucleotide antisensu chì pò hibridà specificamente à a sequenza di destinazione di RNA, à u cuntrariu di primers aleatoriu o oligo (dT), chì anneal à tutti l'RNA.E stesse regule utilizzate per cuncepisce i primeri PCR s'applicanu à u disignu di GSP in reazioni di trascrizione inversa.U GSP pò esse a listessa sequenza cum'è l'amorce di amplificazione chì anneals à l'estremità 3' più di l'ARNm, o u GSP pò esse cuncepitu per anneal downstream di l'amorce di amplificazione inversa.Per certi sughjetti amplificati, più di un primer antisensu deve esse designatu per una RT-PCR riescita perchè a struttura secundaria di l'RNA di destinazione pò impedisce u ligame di u primer.Hè cunsigliatu di utilizà 1 pmol GSP antisensu in una reazione di sintesi di prima fila di 20 μl.
2. Aumentà a temperatura di incubazione per a trascrizione inversa:
Per prufittà di tuttu u vantaghju di a specificità GSP, deve esse aduprata una transcriptase inversa cù una termostabilità più alta.I transcriptasi inversi termostabili ponu esse incubati à temperature più altu per aumentà a stringenza di a reazione.Per esempiu, se un GSP annulla à 55 ° C, a specificità di u GSP ùn serà micca utilizzatu cumplettamente se AMV o M-MLV hè utilizatu per a trascrizione inversa à una bassa stringenza di 37 ° C.In ogni casu, SuperScript II è ThermoScript ponu esse reagiti à 50 ° C o più, chì eliminà i prudutti micca specifichi generati à temperature più bassu.Per a massima specificità, u mischju di RNA/primer pò esse trasferitu direttamente da a temperatura di denaturazione di 65 ° C à a temperatura di incubazione di a trascrizione inversa è aghjunghje à una mistura di reazione 2 × pre-riscaldata (sintesi di cDNA hot start).Questu aiuta à prevene l'accoppiamentu di basa intermolecular à basse temperature.E transizioni multiple di temperatura necessarie per RT-PCR ponu esse simplificate usendu un termociclatore.
3. Reduce a contaminazione di DNA genomicu:
Una difficultà potenziale incontrata cù RT-PCR hè a contaminazione di DNA genomicu in l'RNA.Utilizà un bonu metudu di isolamentu di RNA, cum'è Trizol Reagent, riducerà a quantità di DNA genomicu chì contamina a preparazione di RNA.Per evitari prudutti derivati da DNA genomicu, l'RNA pò esse trattatu cù DNase I di amplificazione per sguassà l'ADN contaminante prima di a trascrizione inversa.A digestione DNase I hè stata terminata incubando i campioni in 2.0 mM EDTA per 10 minuti à 65 ° C.L'EDTA pò chelate l'ioni di magnesiu, prevenendu l'idrolisi di l'RNA dipendente da ioni di magnesiu à alte temperature.
Per separà l'ADNc amplificatu da i prudutti di amplificazione di l'ADN genomicu contaminanti, i primers ponu esse designati chì ogni anneal per separà esoni.I prudutti PCR derivati da cDNA seranu più brevi di quelli derivati da DNA genomicu contaminatu.Inoltre, un esperimentu di cuntrollu senza trascrizzione inversa hè statu realizatu nantu à ogni mudellu di RNA per determinà se un fragmentu determinatu hè derivatu da DNA genomicu o cDNA.U pruduttu PCR ottenutu senza trascrizzione inversa hè derivatu da u genoma.
Tempu di Post: 16-May-2023








