- A PCR hè un metudu utilizatu per amplificà l'ADN da una piccula quantità di mudellu di DNA.RT-PCR usa a trascrizione inversa per pruduce un mudellu di DNA da una fonte di RNA chì pò esse amplificata.
- A PCR è RT-PCR sò tipicamente reazzioni endpoint, mentri qPCR è RT-qPCR utilizanu a cinetica di a tarifa di sintesi di u produttu durante a reazione PCR per quantificà a quantità di mudellu presente.
- I metudi più recenti, cum'è a PCR digitale, furnisce una quantificazione assoluta di u mudellu di DNA iniziale, mentri metudi, cum'è a PCR isotermale, riduce a necessità di equipaggiu caru per furnisce risultati affidabili.
A reazione in catena di polimerasi (PCR) hè una tecnica di biologia moleculare relativamente simplice è largamente usata per amplificà è detectà sequenze di DNA è RNA.Comparatu à i metudi tradiziunali di clonazione è amplificazione di DNA, chì spessu pò piglià ghjorni, a PCR richiede solu uni pochi d'ore.A PCR hè assai sensibile è richiede un mudellu minimu per a rilevazione è l'amplificazione di sequenze specifiche.I metudi di PCR basi anu avanzatu più da a rilevazione simplice di DNA è RNA.Quì sottu, avemu furnitu una panoramica di i diversi metudi di PCR è i reagenti chì furnimu in Enzo Life Sciences per i vostri bisogni di ricerca.Avemu u scopu di aiutà i scientisti à accede rapidamente à i reagenti PCR da aduprà in u so prossimu prughjettu di ricerca!
PCR
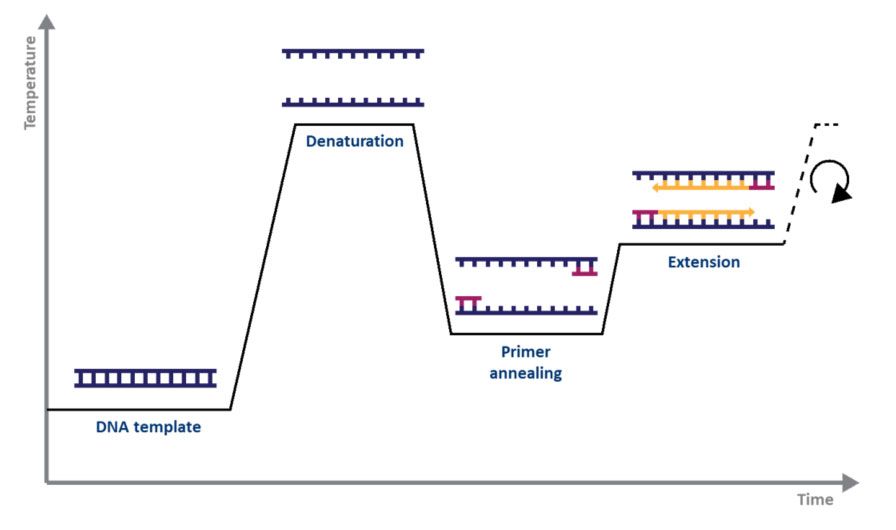
Per a PCR standard, tuttu ciò chì avete bisognu hè una DNA polimerasi, magnesiu, nucleotidi, primers, u mudellu di DNA per esse amplificatu, è un termociclatore.U meccanismo di PCR hè simplice quant'è u so scopu: 1) l'ADN à doppia catena (dsDNA) hè denaturatu da u calore, 2) i primers si allineanu à i filamenti singuli di l'ADN, è 3) i primers sò estesi da l'ADN polimerasi, risultatu in duie copie di l'ADN. filamento di DNA originale.U prucessu di denaturation, annealing, elongation over a series of temperatures and times is known as one cycle of amplification (Fig. 1).
| Figura 1.Rappresentazione schematica di un ciculu di amplificazione per PCR. |
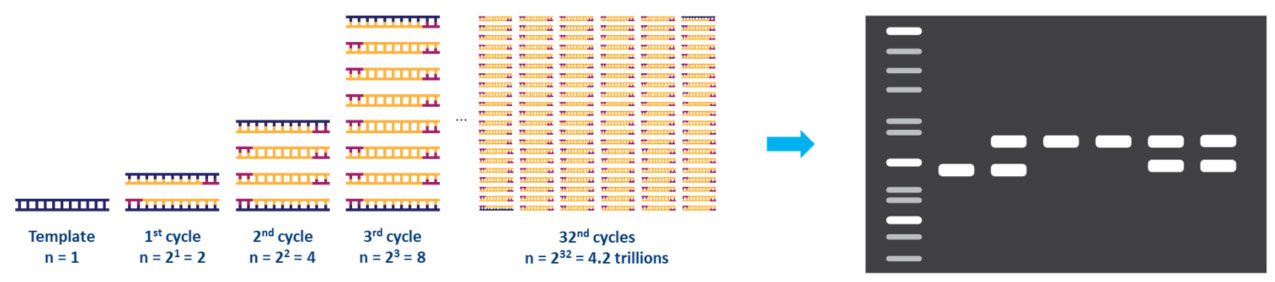
Ogni passu di u ciculu deve esse ottimizatu per u mudellu è u set di primer utilizatu.Stu ciculu hè ripetutu apprussimatamente 20-40 volte, è u pruduttu amplificatu pò esse analizatu, tipicamente da gel d'agarose (Fig. 2).
| Figura 2.Amplificazione di un mudellu di DNA per PCR è analisi per elettroforesi in gel di agarose. |
Siccomu a PCR hè un metudu assai sensitivu è i volumi assai chjuchi sò richiesti per reazzioni singuli, a preparazione di un mischju maestru per parechje reazzioni hè cunsigliatu.U mischju maestru deve esse mischiatu bè è dopu split da u numeru di reazzioni, assicurendu chì ogni reazione cuntene a listessa quantità di enzima, dNTPs è primers.Parechji fornituri, cum'è Enzo Life Sciences, offrenu ancu mischi di PCR chì cuntenenu digià tuttu, eccettu i primers è u mudellu di DNA.
Le regioni ricche di guanina/citosina (ricche di GC) rappresentano una sfida in tecniche standard di PCR.E sequenze ricche di GC sò più stabili di e sequenze cù un cuntenutu GC più bassu.Inoltre, e sequenze ricche di GC tendenu à furmà strutture secondarie, cum'è i loops hairpin.In u risultatu, i filamenti doppiu ricchi di GC sò difficiuli di separà cumplettamente durante a fase di denaturazione.In cunseguenza, l'ADN polimerasi ùn pò micca sintetizà u novu filu senza impedimentu.Una temperatura di denaturazione più alta pò migliurà questu, è l'aghjustamenti versu una temperatura di annealing più altu è un tempu di annealing più brevi ponu impedisce u ligame inspecificu di primers ricchi di GC.Reagenti supplementari ponu rinfurzà l'amplificazione di sequenze ricche di GC.DMSO, glycerol, è betaine aiutanu à disturbà e strutture secondarie chì sò causate da l'interazzione GC è facilitanu cusì a separazione di i filamenti doppiu.
Hot Start PCR
L'amplificazione inspecifica hè un prublema chì pò accade durante a PCR.A maiò parte di l'ADN polimerasi chì sò utilizati per a PCR funziona megliu à a temperatura di circa 68 ° C à 72 ° C.L'enzima pò ancu esse attivu à a temperatura più bassa, ancu s'ellu à un gradu più bassu.À temperatures assai sottu à a temperatura di annealing, i primers ponu ligà in modu micca specificu è portanu à l'amplificazione non specifica, ancu s'è a reazione hè stallata nantu à u ghjacciu.Questu pò esse impeditu cù l'inhibitori di polimerasi chì si dissocianu da l'ADN polimerasi solu una volta chì una certa temperatura hè righjunta, da quì u terminu PCR hot start.L'inhibitore pò esse un anticorpu chì unisce a polimerasi è denatura à a temperatura iniziale di denaturazione (95 ° C tipicamente).
Polimerasi d'alta fedeltà
Mentre l'ADN polimerasi amplificanu abbastanza precisamente à a sequenza di u mudellu originale, ponu accade errori in l'abbinamentu di nucleotidi.Mismatches in appiicazioni cum'è a clonazione pò esse risultatu in trascrizioni truncate, è proteine maltradutte o inattive downstream.Per evitari questi mistches, i polimerasi cù una attività di "lettura di prova" sò stati identificati è incorporati in u flussu di travagliu.A prima polimerasi di correzzione, Pfu, hè stata identificata in u 1991 in Pyrococcus furiosus.Questa enzima Pfu hà un'attività di esonucleasi da 3' à 5'.Quandu l'ADN hè amplificatu, l'esonucleasi elimina i nucleotidi mistched à l'estremità 3' di u filu.U nucleotide currettu hè allora rimpiazzatu, è a sintesi di DNA cuntinueghja.L'identificazione di sequenze di nucleotide incorrecte hè basatu annantu à l'affinità di ubligatoriu per u trifosfatatu di nucleoside currettu cù l'enzima, induve a ligatura inefficiente rallenta a sintesi è permette a sustituzione curretta.L'attività di correzzione di a polimerasi Pfu risulta in menu errori in a sequenza finale paragunata à a polimerasi Taq DNA.Nta l'ultimi anni, altri enzimi di correzione sò stati identificati, è mudificazioni di l'enzima Pfu originale sò state fatte per riducerà ancu a rata di errore durante l'amplificazione di DNA.
RT-PCR
A PCR di trascrizzione inversa, o RT-PCR, permette l'usu di RNA cum'è mudellu.Un passu supplementu permette a deteczione è l'amplificazione di l'RNA.L'RNA hè trascrittu inversamente in DNA cumplementariu (cDNA), utilizendu a transcriptase inversa.A qualità è a purità di u mudellu RNA sò essenziali per u successu di RT-PCR.U primu passu di RT-PCR hè a sintesi di un hibridu DNA / RNA.A transcriptase inversa hà ancu una funzione RNase H, chì degrada a parte RNA di l'ibridu.A molècula di DNA monofila hè allora cumpletata da l'attività di DNA polimerasi dipendente da l'ADN di a transcriptase inversa in cDNA.L'efficienza di a reazione di u primu filu pò influenzà u prucessu di amplificazione.Da quì in avanti, a prucedura standard di PCR hè aduprata per amplificà u cDNA.A pussibilità di rinvià l'RNA in cDNA da RT-PCR hà assai vantaghji, è hè principalmente utilizatu per l'analisi di l'espressione genica.L'RNA hè unicu filatu è assai inestabile, chì face sfida à travaglià.Hè comunmente serve cum'è un primu passu in qPCR, chì quantifica transcripts di RNA in una mostra biologica.
qPCR è RT-qPCR
A PCR quantitativa (qPCR) hè aduprata per detectà, caratterizà è quantificà l'acidi nucleici per numerose applicazioni.In RT-qPCR, i trascrizioni di RNA sò spessu quantificate da trascrizzione inversa in cDNA prima, cum'è descrittu sopra, è dopu qPCR hè susseguitu.Cum'è in a PCR standard, l'ADN hè amplificatu da trè passi ripetuti: denaturazione, annealing è allungamentu.Tuttavia, in qPCR, l'etichettatura fluorescente permette a cullizzioni di dati mentre a PCR avanza.Sta tecnica hà assai benefici per via di a gamma di metudi è chimichi dispunibili.
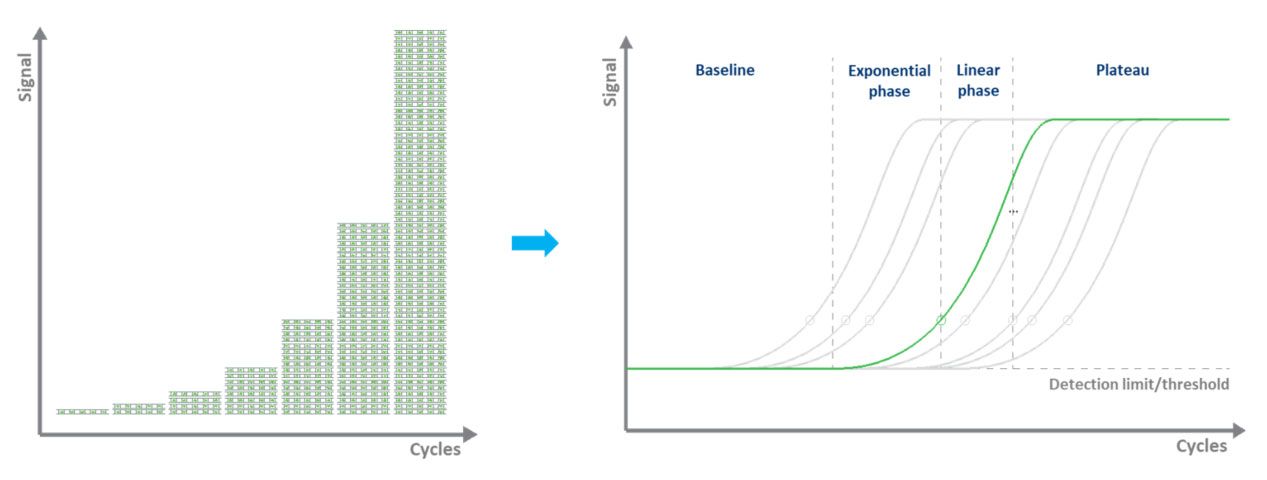
In qPCR basata in tintura (tipicamente verde), l'etichettatura fluorescente permette a quantificazione di e molécule di DNA amplificate utilizendu l'usu di un colorante di legame dsDNA.Durante ogni ciculu, a fluoriscenza hè misurata.U signale di fluorescenza aumenta proporzionalmente à a quantità di DNA replicatu.Dunque, l'ADN hè quantificatu in "tempu reale" (Fig. 3).I svantaghji di a qPCR basata in tintura sò chì solu un mira pò esse esaminatu à u mumentu è chì u tintu si unisce à qualsiasi ds-DNA presente in a mostra.
| Figura 3.Amplificazione di un mudellu di DNA da qPCR è misurazione di u signale di fluorescenza in tempu reale. |
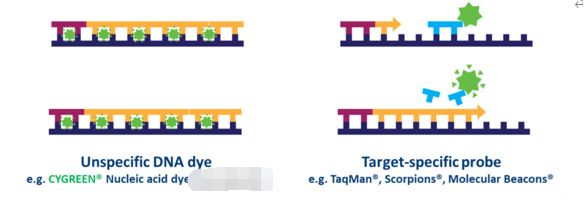
In a qPCR basata in sonda, parechji miri ponu esse rilevati simultaneamente in ogni mostra, ma questu richiede l'ottimisazione è u disignu di una sonda (s) specifica per u target utilizata in più di i primers.Diversi tipi di disinni di sonda sò dispunibuli, ma u tipu più cumuni hè una sonda d'idrolisi, chì incorpora un fluoroforu è quencher.U trasferimentu di energia di risonanza di fluorescenza (FRET) impedisce l'emissione di u fluoroforu via u quencher mentre a sonda hè intacta.Tuttavia, durante a reazione di PCR, a sonda hè idrolizzata durante l'estensione di l'amori è l'amplificazione di a sequenza specifica à a quale hè ligata.A clivatura di a sonda separa u fluoroforu da u quencher è risultati in un incrementu di fluoriscenza dipendente da l'amplificazione (Fig. 4).Cusì, u signale di fluorescenza da una reazione qPCR basata in una sonda hè proporzionale à a quantità di a sequenza di destinazione di a sonda presente in a mostra.Perchè a qPCR basata in sonda hè più specifica di a qPCR basata in tintura, hè spessu a tecnulugia aduprata in test di diagnostichi basati in qPCR.
| Figura 4.Differenze trà qPCR basati in tintura è basati in sonda. |
Amplificazione isotermica
I tecnichi di PCR sopra citati necessitanu un equipaggiu di termociclamentu caru per accurtà accuratamente a temperatura di a camera per i passi di denaturazione, annealing è estensione.Una quantità di tecnichi sò stati sviluppati chì ùn anu micca bisognu di tali dispusitivi precisi è ponu esse realizati in un bagnu d'acqua simplice o ancu in e cellule d'interessu.Sti tecnichi sò cullettivamente chjamati amplificazione isotermica è u travagliu basatu annantu à l'amplificazione esponenziale, lineale o in cascata.
U tipu più cunnisciutu di amplificazione isotermica hè l'amplificazione isotermica mediata da loop, o LAMP.LAMP usa l'amplificazione esponenziale à 65⁰C per amplificà l'ADN o l'RNA mudellu.Quandu eseguisce LAMP, quattru à sei primers cumplementarii à e regioni di l'ADN di destinazione sò usati cù una DNA polimerasi per sintetizà novu DNA.Dui di questi primers anu sequenze cumplementarii chì ricunnoscenu e sequenze in l'altri primers è li liganu, chì permettenu una struttura di "ciclu" per furmà in l'ADN di sintesi di novu chì aiuta à l'annealing di primer in i turni successivi di amplificazione.LAMP pò esse visualizatu da parechji metudi, cumprese fluorescenza, elettroforesi in gel d'agarose, o colorimetria.A facilità di visualizà è detectà a prisenza o l'assenza di produttu per colorimetria è a mancanza di l'equipaggiu caru necessariu hà fattu di LAMP una opzione adatta per a prova SARS-CoV-2 in i zoni induve e teste di laboratori clinichi ùn eranu micca prontamente dispunibili, o u almacenamentu è u trasportu di campioni. ùn era micca fattibile, o in laboratori chì ùn anu micca avutu l'equipaggiu di termociclamentu PCR.
Tempu di post: Aug-19-2023