Materiale di partenza: RNA
A PCR di trascrizione inversa quantitativa (RT-qPCR) hè un metudu sperimentale utilizatu in esperimenti di PCR chì utilizanu RNA cum'è materiale di partenza.In questu metudu, l'RNA tutale o l'RNA messenger (mRNA) hè prima trascrittu in DNA cumplementariu (cDNA) da a transcriptase inversa.In seguitu, una reazione qPCR hè stata realizata utilizendu u cDNA cum'è mudellu.RT-qPCR hè stata utilizata in una varietà di applicazioni di biologia moleculare, cumprese l'analisi di l'espressione genica, a validazione di l'interferenza di RNA, a validazione di microarray, a rilevazione di patogeni, i testi genetichi è a ricerca di e malatie.
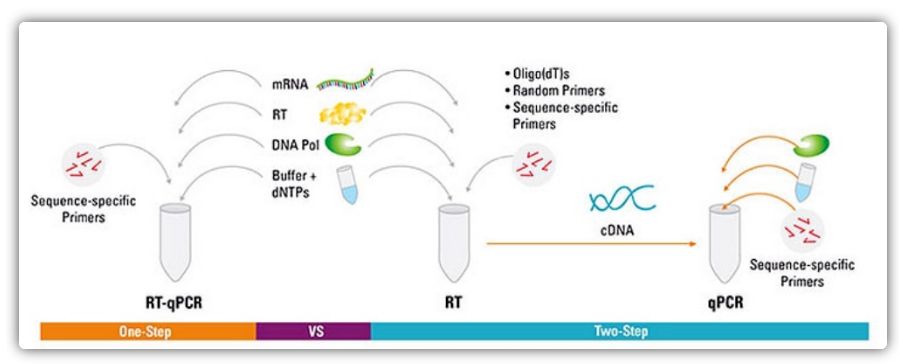
Metodi à un passu è dui passi per RT-qPCR
RT-qPCR pò esse realizatu da un metudu in un passu o dui passi.L'RT-qPCR in un passu combina a trascrizione inversa è l'amplificazione PCR, chì permette a transcriptase inversa è l'ADN polimerasi per cumpletà a reazione in u stessu tubu in e stesse cundizioni di buffer.L'RT-qPCR in un passu richiede solu l'usu di primers specifichi di sequenza.In RT-qPCR in dui tappe, a trascrizione inversa è l'amplificazione PCR sò realizate in dui tubi, utilizendu diversi buffer ottimizzati, cundizioni di reazione è strategie di cuncepimentu di primer.
| Vantage | Svantaghju | |
| Un Passu | Stu metudu hà menu errore spirimintali chì e duie reazzioni sò fatti in un tubu
Meno passi di pipetting riduce u risicu di contaminazione
Adatta per amplificazione / screening high-throughput, veloce è riproducibile | E reazzioni in dui passi ùn ponu esse ottimizzati separatamente
Siccomu e cundizioni di reazione sò cumprumessi cumminendu a reazione in dui passi, a sensibilità ùn hè micca cusì bona cum'è quella di u metudu in dui passi.
U nùmeru di mira rilevati da una sola mostra hè chjuca |
| Dui Passi | Capacità di creà biblioteche cDNA stabili chì ponu esse almacenati per longu periudi di tempu è utilizati in parechje reazioni
I geni di destinazione è i geni di riferimentu ponu esse amplificati da a stessa biblioteca di cDNA senza a necessità di parechje biblioteche di cDNA
Buffer di reazione è e cundizioni di reazione chì permettenu l'ottimisazione di una sola corsa di reazione
Selezzione flessibile di e cundizioni di trigger | L'usu di più tubi, è più passi di pipetting aumenta u risicu di contaminazione di DNA, è cunsumanu tempu.
Esige più ottimisazione cà u metudu in un passu |
I prudutti cunnessi:
RT-qPCR Easyᵀᴹ (Un Passu)-SYBR Green I
RT-qPCR Easyᵀᴹ (Un passu)-Taqman
RT Easyᵀᴹ I Master Premix per a sintesi CDNA di u primu filu
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
Selezzione di RNA totale è mRNA
Quandu si cuncepisce un esperimentu RT-qPCR, hè impurtante decide di utilizà RNA tutale o mRNA purificatu cum'è mudellu per a trascrizione inversa.Ancu s'ellu mRNA pò esse capaci di furnisce una sensibilità ligeramente più altu, l'RNA tutale hè sempre spessu usatu.U mutivu di questu hè chì l'RNA tutale hà un vantaghju più impurtante cum'è materiale di partenza chì mRNA.Prima, u prucessu richiede menu passi di purificazione, chì assicura una megliu ricuperazione quantitativa di u mudellu è una megliu normalizazione di i risultati à i numeri di cellule di partenza.Siconda, evita u passu di arricchimentu di mRNA, chì pò evità a pussibilità di risultati distorti per via di e diverse ricuperazioni di diversi mRNA.In generale, postu chì in a maiò parte di l'applicazioni a quantificazione relativa di u genu di destinazione hè più impurtante chè a sensibilità assoluta di a deteczione, l'RNA tutale hè più adattatu in a maiò parte di i casi.
Primer di trascrizione inversa
In u metudu di dui passi, trè metudi diffirenti ponu esse utilizati per prima a reazione di cDNA: primers oligo (dT), primers aleatoriu, o primers specifichi di sequenza.Di genere, oligo (dT) primers è primers aleatori sò usati in cumminazione.Questi primers anneanu à u filu di mRNA di u mudellu è furnisce a transcriptase inversa cun un puntu di partenza per a sintesi.
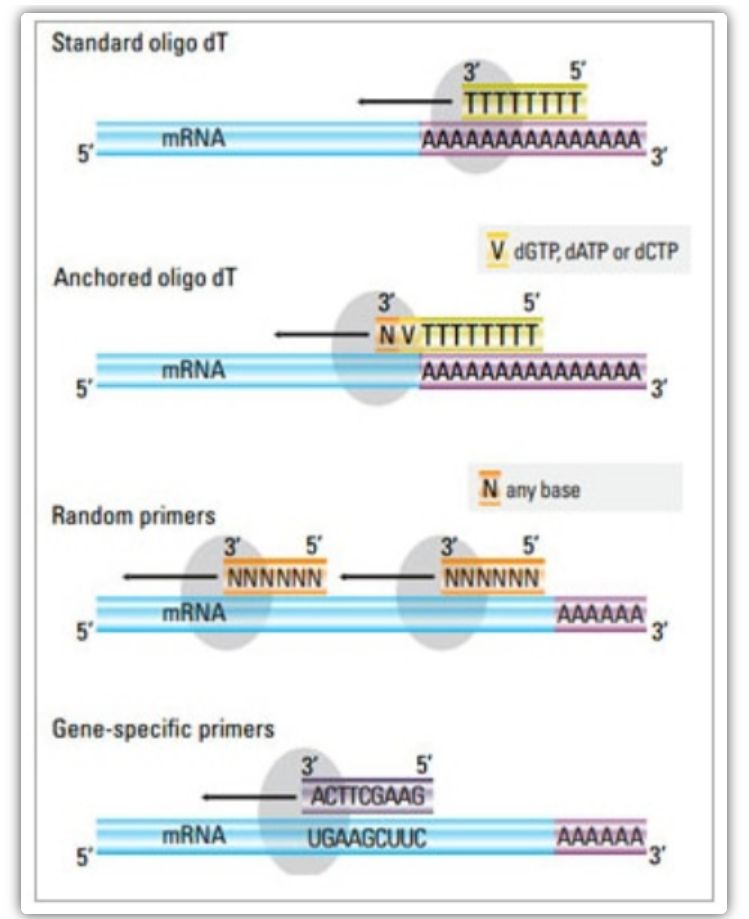
| Selezzione di prima | Struttura è funzione | Vantage | Svantaghju |
| Primer oligo(dT) (o primer oligo(dT) ancorato) | Annealing estesa à i residui di timina à a coda poli (A) di mRNA;L'amorce d'oligo(dT) d'ancora cuntene un G, C, o A à l'estremità 3' (situ di ancora) | Sintesi di cDNA full-length da mRNA poly(A)-tailed
Applicabile quandu menu materiale di partenza hè dispunibule
Le site d'ancrage garantit que l'amorce oligo(dT) se lie à la queue poly(A) 5' de l'ARNm | Solu adattatu per amplificazione di geni cù code poly(A).
Ottene cDNA truncatu da u situ di primura * 2 in poly(A)
Biased per ligà à l'estremità 3 '*
*Sta pussibilità hè minimizzata se sò usati primers oligo(dT) ancorati |
| primer casuale
| 6 à 9 basi in lunghezza, chì ponu anneling à parechji siti durante a trascrizione di RNA | Anneal à tutti l'RNA (tRNA, rRNA è mRNA)
Adatta per trascrizioni cù una struttura secundaria significativa, o quandu menu materiale di partenza hè dispunibule
Altu rendimentu di cDNA | L'ADNc hè trascrittu inversamente da tutti l'ARN, chì generalmente ùn hè micca desideratu è pò diluisce u signale di l'ARNm di destinazione.
ottene cDNA truncatu |
| primers specifici di sequenza | Primer personalizzati destinati a sequenze di mRNA specifiche | libreria cDNA specifica
Migliurà a sensibilità
Utilizendu i primeri qPCR inversi | Solu limitatu à a sintesi di un unicu genu di destinazione |
Trascrittasi inversa
A transcriptase inversa hè una enzima chì usa RNA per sintetizà DNA.Alcune transcriptasi inverse anu attività di RNase è ponu degradare i filamenti di RNA in filamenti hibridi RNA-DNA dopu a trascrizione.Se ùn hà micca attività enzimatica RNase, RNaseH pò esse aghjuntu per una più alta efficienza qPCR.L'enzimi cumunimenti usati includenu a transcriptase inversa di u virus di leucemia murina Moloney è a transcriptase inversa di u virus mieloblastoma aviari.Per RT-qPCR, hè ideale per sceglie una transcriptase inversa cù una termostabilità più alta, perchè a sintesi di cDNA pò esse realizata à temperature più elevate, assicurendu una trascrizzione riescita di RNAs cù una struttura secundaria più alta, mantenendu a so attività completa in tutta a reazione, risultatu in un rendimentu di cDNA più altu.
I prudutti cunnessi:
Trascrittasi inversa di Foreasy M-MLV
Attività RNase H di trascrittasi inversa
RNaseH hè capaci di degradare i filamenti di RNA da i duplexi RNA-DNA, permettendu una sintesi efficiente di DNA a doppia catena.In ogni casu, quandu si usa l'ARNm longu cum'è mudellu, l'ARN pò esse degradatu prematuramente, risultatu in un ADNc truncatu.Per quessa, hè spessu benefiziu per minimizzà l'attività di RNaseH durante a clonazione di cDNA se a sintesi di trascrizioni longu hè desiderata.In cuntrastu, i transcriptasi inversi cù l'attività RNase H sò spessu benefizii per l'applicazioni qPCR perchè aumentanu a fusione di duplex RNA-DNA durante u primu ciclu di PCR.
Disegnu di prima
I primers di PCR usati per u passu qPCR in RT-qPCR sò idealmente pensati per abbracciare una giunzione esone-esone, induve un primer di amplificazione puderia potenzialmente span un cunfine esone-introne attuale.Siccomu e sequenze di DNA genomicu chì cuntenenu introni ùn sò micca amplificate, stu disignu reduce u risicu di falsi pusitivi amplificati da a contaminazione di DNA genomicu.
Se i primers ùn ponu micca esse designati per separà esoni o frontiere esone-esone, pò esse necessariu di trattà campioni di RNA cù DNase I o dsDNase senza RNasi per sguassà a contaminazione di DNA genomicu.
cuntrollu RT-qPCR
Un cuntrollu negativu di trascrizione inversa (controllo -RT) deve esse inclusu in tutti l'esperimenti RT-qPCR per detectà a contaminazione di l'ADN (cum'è DNA genomicu o prudutti di PCR da reazzione precedente).Stu cuntrollu cuntene tutti i cumpunenti di reazzione eccettu a transcriptase inversa.Siccomu a trascrizione inversa ùn si trova micca cù questu cuntrollu, se l'amplificazione di PCR hè osservata, a contaminazione da DNA hè più prubabile.
Tempu di Postu: Aug-02-2022








